Publicado em junho 22, 2022 por glaucepereira
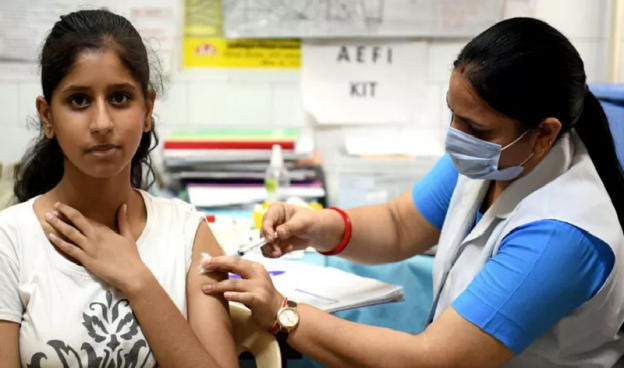
Um estudo conduzido pelo Instituto Sabin de Vacinas e pela Fiocruz Mato Grosso do Sul vai avaliar a possibilidade de fracionar as doses de reforço dos imunizantes para a Covid-19. A pesquisa deverá mostrar se doses menores podem oferecer a mesma resposta imunológica e com reações adversas menores. Isso possibilitaria multiplicar a oferta de vacinas, principalmente nos países mais pobres, e orientar novas estratégias de imunização. Segundo o Instituto Sabin, apenas 17,4% da população dos países de baixa renda foram vacinados contra a Covid-19, enquanto nos países alta renda esse índice chega a 72%.
Para o estudo, o instituto recebeu US$ 6,3 milhões (R$ 32,7 milhões) da Coalizão para Promoção de Inovações em prol da Preparação para Epidemias (Cepi, a sigla em inglês), uma organização internacional que tem como objetivo financiar projetos de pesquisa para acelerar a produção de imunizantes. O instituto escolheu, então, dois países: Brasil e Paquistão. Em cada um deles, 1.440 pessoas participarão da pesquisa, recebendo as vacinas Pfizer (dose cheia, metade ou um terço), AstraZeneca (dose cheia ou meia) e Coronavac (dose cheia), sendo acompanhadas por seis meses.
Leia Mais