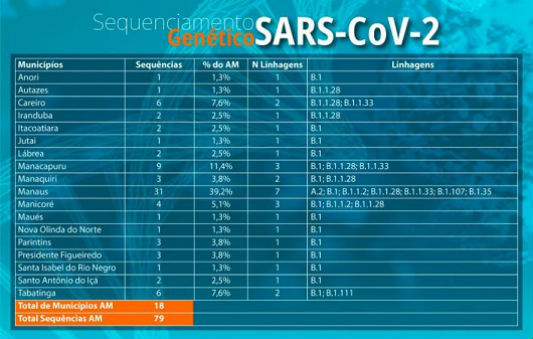
Após nove meses do surgimento do primeiro caso de Covid-19 no Amazonas, pesquisadores do Instituto Leônidas & Maria Deane (ILMD/Fiocruz Amazônia) já sequenciaram 79 genomas do Sars-CoV-2, a partir de amostras obtidas em 18 municípios do estado. A pesquisa contou com a parceria da Fundação de Vigilância em Saúde do Amazonas (FVS/AM), por meio do Laboratório Central de Saúde Pública (Lacen-AM), dentro de uma estratégia de vigilância de vírus emergentes/reemergentes e negligenciados, iniciada há mais de cinco anos.
Liderado pelo pesquisador e vice-diretor de Pesquisa e Inovação do ILMD/Fiocruz Amazônia, Felipe Naveca, o estudo encontrou oito linhagens do novo coronavírus circulando no estado, o que sugere ao menos oito introduções do Sars-CoV-2 no Amazonas. Outra importante descoberta foi a identificação de quatro linhagens que ainda não haviam sido sequenciadas no Brasil.
As quatro novas linhagens identificadas são a B.1.107; B.1.111; B.1.1.2; e B.1.35 que circularam na Dinamarca, Colômbia, Reino Unido e País de Gales, respectivamente. Com essas, sobem para 30 o número de linhagens encontradas no Brasil. “Quanto mais nós investigamos, maior variabilidade temos encontrado, o que nos mostra que foram diversas as entradas do vírus no estado, mesmo em um período onde, teoricamente, havia menor circulação de pessoas”, comenta Naveca.
As amostras sequenciadas no ILMD/Fiocruz Amazônia são provenientes dos municípios de Anori, Autazes, Careiro, Iranduba, Itacoatiara, Jutaí, Lábrea, Manacapuru, Manaquiri, Manicoré, Maués, Nova Olinda do Norte, Parintins, Presidente Figueiredo, Santa Isabel do Rio Negro, Santo Antônio do Içá, e Tabatinga, além da capital Manaus.
O pesquisador reforça a importância do sequenciamento do novo coronavírus e a continuidade desse trabalho, inclusive para os protocolos de diagnóstico. “Os dados do sequenciamento nos ajudam também a verificar se há a necessidade de ajustes nos protocolos de diagnóstico, por exemplo, se aqui o vírus acumulou alguma mutação que leve a um resultado falso-negativo. Os protocolos em uso hoje foram desenvolvidos em outros países, como China, EUA e Alemanha, levando em consideração o que se sabia da variabilidade viral naquele momento”, afirma.
Para o diretor-técnico FVS-AM, Cristiano Fernandes, novos questionamentos surgem com a identificação das linhagens do Sars-CoV-2 que circulam no Amazonas, especialmente sobre o percurso do vírus até ser introduzido nos municípios. “O nosso desafio é ampliar as amostras dos municípios do interior para tentar identificar maior variabilidade viral, entendendo o contexto e a complexidade que a doença tem nos trazido, lembrando que são quase 4.600 óbitos aqui no estado, então, algumas perguntas ainda precisam ser respondidas e esse estudo pode esclarecer muito do ponto de vista da vigilância, esse aspecto relacionado à patologia da doença, baseado em estudos genômicos e de variabilidade viral”, declara Fernandes.
Imagem: Mackesy Nascimento
Genômica no Amazonas
Em março deste ano, a poucos dias da ocorrência do primeiro caso de Covid-19 no Amazonas, Felipe Naveca concluiu o primeiro sequenciamento do genoma do Sars-CoV-2 do Norte do país. O estudo é continuo, dada a importância de se entender melhor a dispersão do vírus no Amazonas. Vale ressaltar que o sequenciamento dos genomas de Sars-CoV-2 contribuem para o desenvolvimento de vacinas e medicamentos contra o vírus. Os genomas identificados no Amazonas integram um banco de dados e podem ser comparados a outros que circulam no Brasil e no mundo.
O estudo de Felipe Naveca e sua equipe é apoiado pela Fundação de Amparo à Pesquisa do Estado do Amazonas (Fapeam), por meio da Rede Genômica de Vigilância em Saúde do Estado do Amazonas (Regesam) e do edital PCTI-EmergeSaúde AM; pelo Conselho Nacional de Desenvolvimento Científico e Tecnológico (CNPq), através da Chamada 07/2020 – Pesquisas para enfrentamento da Covid-19; e pela Fiocruz, edital Inova – Geração de conhecimentos.