Em parceria com o Instituto Leônidas e Maria Deane (ILMD / Fiocruz Amazônia), pesquisadores da Fiocruz Rondônia identificaram três variantes do vírus Sars-CoV-2, circulantes no estado (RO). Os dados foram apresentados durante transmissão realizada pelo Governo nesta terça-feira (9/2). As análises foram realizadas com base em amostras de pacientes coletadas em diferentes municípios, incluindo a capital (Porto Velho). O estudo contou com colaboração da Secretaria de Estado da Saúde (Sesau), do Laboratório Central de Saúde Pública (Lacen) e do Centro de Pesquisa em Medicina Tropical (Cepem).
De acordo com a virologista e pesquisadora em Saúde Pública da Fiocruz Rondônia, Deusilene Vieira, desde que começaram os estudos para o acompanhamento e vigilância genômica do vírus, foram descritas pela comunidade científica diversas variantes, observadas em todo o mundo. “A cada mudança do vírus, são geradas também novas linhagens, o que justifica a necessidade de novos estudos para uma melhor compreensão dos fatores clínicos e epidemiológicos relacionados à doença”, enfatizou Deusilene Vieira.
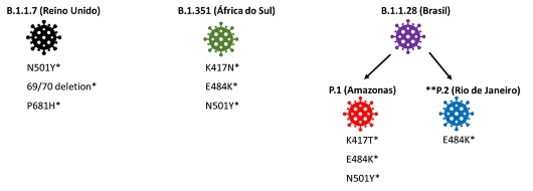
A partir de isolados virais de dezembro de 2020 e janeiro de 2021, a vigilância genômica de Sars-CoV-2 em Rondônia permitiu identificar as seguintes linhagens do vírus: B.1.1.33 (já identificada em todos os continentes, sendo comum na América do Sul); B.1.1.28 (comum no Brasil e já localizada em outros países) e P.2 (variante que foi descrita primeiramente no Rio de Janeiro, e já identificada também em outras localidades do Brasil).
A pesquisadora ressaltou a importância de todos seguirem as medidas restritivas. “Isso além do uso de máscara, distanciamento social e álcool em gel para evitar a propagação do vírus, “uma vez que os dados dessas análises referentes aos meses de dezembro/2020 e janeiro/2021 demonstram maior transmissibilidade do vírus, o que também está relacionado a mutações detectadas nessas variantes”, concluiu Vieira.
Médico infectologista, Juan Miguel Villalobos-Salcedo pontuou que não se pode afirmar que as novas variantes, identificadas em Rondônia, são mais agressivas e estão relacionadas ao aumento considerável no número de mortes, verificado no estado a partir do último semestre do ano passado. Ele explicou que a principal característica encontrada, a partir das análises realizadas, é o alto potencial de transmissão do vírus, inclusive entre os mais jovens. “Quanto mais casos ativos tivermos em uma população, mais chances nós temos de propagar a doença e, consequentemente, mais possibilidades teremos de provocar novas mutações do vírus”, destacou.
Mutações
Os vírus vão adquirindo, normalmente, mudanças em seu genoma e estas se acumulam conforme o tempo progride. Essas mudanças, também chamadas de mutações, podem ser utilizadas para ler e inferir o passado evolutivo de um vírus, baseando-se no fato de que fica registrada em seu genoma toda a rota mutacional que levou à sua geração.
Conforme esse vírus se espalha e infecta novas pessoas, o vírus vai acumulando novas mutações, pois as cepas vão percorrendo caminhos e ambientes distintos. As diferentes pressões como o sistema imunológico e fatores ambientais se encarregam de selecionar quais “cepas” (variantes) seguirão adiante, repassando informações genéticas, durante as infecções.
Estes caminhos diferentes vão dando origem a diferentes linhagens de um vírus. Linhagem, portanto, pode ser entendida como um grupo de vírus que compartilham uma série de gerações ou linha de parentesco e são, portanto, semelhantes geneticamente entre si. Linhagens específicas surgem a partir do aparecimento de variantes virais novas, que normalmente apresentam uma ou mais mutações diferentes das demais variantes circulantes em uma população, e que conseguem passar essas informações por várias gerações adiante. Assim sendo, por vezes, novas variantes surgem e desaparecem, enquanto outras novas variantes podem surgir e persistir.
Atualmente, em nível global, três linhagens de Sars-CoV-2 são consideradas emergentes e de grande importância epidemiológica: B.1.1.7 (originária do Reino Unido); B.1.351 (da África do Sul) e P1 ou B.1.1.28.1 (identificada no Brasil/Amazonas). Todas estas linhagens têm sido associadas a uma maior transmissibilidade do vírus. No Brasil, emergiu uma linhagem que se espalhou para alguns outros continentes do mundo e foi identificada como B.1.1.28. Descendendo desta linhagem, outras duas surgiram: P.1, a linhagem que surgiu no Estado do Amazonas e a P.2, uma variante isolada primeiramente no Rio de Janeiro e que já está presente em outras localidades do Brasil.