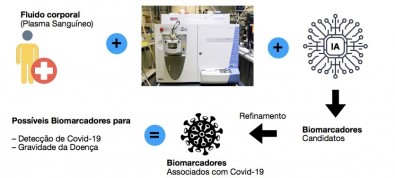
Sistema automatizado desenvolvido por pesquisadores da Unicamp, da USP e colaboradores se baseia na análise do padrão de moléculas do plasma sanguíneo de pacientes.
Um método que permite diagnosticar a COVID-19 em cerca de 20 minutos – com baixo custo e sem a necessidade de reagentes importados – foi descrito por pesquisadores brasileiros em artigo divulgado na plataforma medRxiv, ainda sem revisão por pares.
O sistema usa algoritmos de inteligência artificial capazes de reconhecer em amostras de plasma sanguíneo de pacientes um padrão de moléculas característico da doença. Segundo os autores, também é possível identificar, entre os casos confirmados, os indivíduos com maior risco de desenvolver manifestações graves, como insuficiência respiratória.
O projeto é apoiado pela FAPESP e envolve pesquisadores da Universidade Estadual de Campinas (Unicamp) e da Universidade de São Paulo (USP), além de colaboradores no Amazonas.
“Nos testes feitos para validar a metodologia, conseguimos diferenciar as amostras positivas e negativas com um acerto de mais de 90%. Também fizemos a diferenciação entre casos graves e leves com acerto em torno de 82%. Agora, estamos iniciando o processo de certificação junto à Anvisa [Agência Nacional de Vigilância Sanitária]”, conta à Agência FAPESP o professor da Unicamp Rodrigo Ramos Catharino, coordenador da pesquisa.
Segundo ele, o exame, quando em operação, poderia custar em torno de R$ 40 por amostra, cerca de metade do preço de custo do RT-PCR, método considerado padrão-ouro para diagnóstico da COVID-19.
O trabalho vem sendo desenvolvido no Laboratório Innovare de Biomarcadores, durante o doutorado de Jeany Delafiori, e integra uma linha de pesquisa que combina técnicas de metabolômica e aprendizado de máquina para buscar marcadores capazes de auxiliar o diagnóstico de doenças como zika, dengue hemorrágica, fibrose cística, diabetes e outros distúrbios metabólicos.
O grupo trabalha em parceria com o Laboratório de Inferência em Dados Complexos (Recod) do Instituto de Computação (IC) da Unicamp, coordenado pelo professor Anderson Rocha e conta com a participação de seu colaborador Luiz Claudio Navarro.
“O projeto contou com a participação de 728 pacientes – sendo 369 com diagnóstico de COVID-19 confirmado clinicamente e por RT-PCR. As amostras de indivíduos não infectados foram usadas para comparação, como uma espécie de grupo controle. No caso de alguns pacientes que desenvolveram complicações e precisaram ser internados, foi coletada uma segunda amostra de sangue. De modo geral, entre os casos confirmados, havia indivíduos com sintomas leves e graves”, conta Delafiori.
Todas as amostras foram analisadas em um equipamento conhecido como espectrômetro de massas, capaz de discriminar as substâncias presentes em fluidos corporais. Como explicam os pesquisadores, esse conjunto de moléculas encontrado no plasma sanguíneo retrata os diversos processos metabólicos ativos no organismo.
“Nos concentramos nas moléculas de baixo peso molecular, como os aminoácidos, os pequenos peptídeos e os lipídeos. Elas surgem na parte final dos processos metabólicos e, portanto, estão mais diretamente ligadas aos sintomas que os pacientes estavam manifestando no momento da coleta”, explica Delafiori.
Parte das amostras foi então usada pela equipe do IC-Unicamp para ensinar um método de inteligência artificial a reconhecer padrões de metabólitos encontrados nos casos positivos e nos negativos, bem como diferenciar os padrões dos casos leves e graves. A outra parte foi usada em um teste cego, cujo objetivo foi avaliar o acerto final da análise feita pelo sistema.
Segundo os dados do artigo, o método alcançou 97,6% de especificidade e 83,8% de sensibilidade para o diagnóstico da doença no teste cego. Já em relação à análise de risco para manifestação grave a especificidade foi de 76,2% e a sensibilidade foi de 87,2%.
“Sensibilidade [também conhecido como sensitividade] é o parâmetro que indica o quão sensível o método é para detectar a presença ou ausência da COVID-19. Já especificidade tem a ver com a capacidade de diferenciar a COVID-19 de outras condições de saúde. Esses dois parâmetros, quando analisados em conjunto, determinam a taxa de acerto”, explica Delafiori. “Ainda estamos trabalhando para melhorar a taxa de acerto do exame, à medida que novas amostras de pacientes são coletadas por nossos colaboradores.”
De acordo com Rocha, o algoritmo desenvolvido é capaz de incorporar conhecimento na medida em que analisa novas amostras, o que tende a se refletir em uma melhora na performance com o passar do tempo. “Se hoje ele tem uma taxa de acerto em torno de 90%, é provável que acerte ainda mais quando chegar a milhares de pacientes analisados”, afirma o pesquisador.
A equipe do IC-Unicamp também criou um software para automatizar todo o processo de análise e gerar, no final, um relatório que informa para o médico se o paciente tem a COVID-19 e se apresenta risco de complicações.
“Esses biomarcadores preditores de evolução da doença podem, por exemplo, ajudar o médico da assistência básica a decidir se o paciente que testar positivo pode ser mantido em isolamento domiciliar ou se deve ser transferido para um centro de maior complexidade”, comenta Rinaldo Focaccia Siciliano, médico assistente da Divisão de Moléstias Infecciosas e Parasitárias do Hospital das Clínicas (HC-FM-USP) e da Unidade de Controle de Infecção Hospitalar do Instituto do Coração (InCor), um dos coautores do artigo.
Na avaliação de Siciliano, o método tem mostrado um bom desempenho para detectar tanto os casos leves, nos primeiros dias de sintomas, como também os mais avançados, de pacientes que já apresentam falta de ar na admissão ao hospital. “A vantagem de ter vários centros participando do projeto, com diferentes perfis, é a variabilidade das amostras. Isso permite que seja possível aplicar a metodologia em diferentes cenários, tanto ambulatorial quanto hospitalar”, diz.
Outro avanço apontado pelo pesquisador é a possibilidade de diagnosticar precocemente a doença por meio de uma amostra de sangue, mais fácil de ser coletada do que a secreção nasal usada no teste de RT-PCR. “A coleta com swab [cotonete comprido inserido no fundo do nariz] requer equipe bem treinada e sala apropriada, pois há risco de dispersão de aerossóis contaminados com o vírus. E o teste sanguíneo atualmente disponível só é capaz de detectar anticorpos alguns dias depois do surgimento dos sintomas.”
Modelo otimizado
Enquanto a maioria dos testes laboratoriais analisa no sangue os níveis de algumas poucas substâncias, o sistema computacional desenvolvido pela equipe da Unicamp é capaz de olhar, ao mesmo tempo, para milhares de variáveis e extrair interconexões diretas e cruzadas entre elas: por exemplo, quais substâncias estão aumentadas e quais estão diminuídas em indivíduos com uma determinada doença.
“Para tornar isso possível, trabalhamos nos últimos três anos no desenvolvimento de um modelo matemático que seja explicável, ou seja, que nos permita não apenas fazer uma predição correta como também saber quais variáveis o sistema está olhando para fazer essa predição. Isso possibilita, após a identificação de um primeiro conjunto de biomarcadores, selecionar os mais significativos e otimizar o processo de análise. Além disso, os dados gerados podem ser usados pela área de metabolômica para desvendar o mecanismo da doença”, explica Navarro.
No caso da COVID-19, o grupo chegou a um conjunto de aproximadamente 30 metabólitos que funcionam como uma assinatura da doença. De acordo com Delafiori, o diagnóstico positivo foi associado, por exemplo, a uma redução no nível de lisofosfatidilcolinas – fosfolipídios derivados de glicerol que contêm fosfato na sua estrutura. “Essas moléculas são precursoras de surfactantes pulmonares [compostos que reduzem a tensão superficial dentro do alvéolo pulmonar, prevenindo o colapso durante a expiração] e protegem o órgão de infecções oportunistas. A diminuição dessas espécies foi reportada previamente em pacientes com síndrome respiratória aguda grave”, conta.
Também se observou nos casos positivos, diminuição nos derivados de colesterol, que foi ainda mais pronunciada nos pacientes que evoluíram para a forma grave. “Alguns estudos reportam redução nos níveis de colesterol à medida que o paciente com COVID-19 evolui para um desfecho negativo”, diz a pesquisadora.
Já os níveis de glicerolpídios – reportados previamente desregulados na síndrome respiratória aguda grave – estavam aumentados nas amostras de pacientes com a doença.
“Após essa etapa de validação bioquímica dos biomarcadores – que permitiu, por exemplo, descartar moléculas associadas ao uso de um medicamento anti-inflamatório que não tinham relação com a doença – combinamos as variáveis restantes em pares. Essa nova técnica que estamos introduzindo no modelo aumenta a acurácia da análise e a torna possível de ser feita com diferentes equipamentos de espectrometria de massas”, conta Navarro.
Na avaliação de Catharino, a metodologia poderia ser aplicada em qualquer laboratório público ou privado equipado com um espectrômetro de massas. Enquanto protocolam o registro na Anvisa, os pesquisadores pretendem aumentar ainda mais a diversidade das amostras analisadas no contexto da pesquisa para melhorar a performance do sistema.
O grupo conta com a colaboração de pesquisadores da Universidade do Estado do Amazonas (UEA), da Fundação de Medicina Tropical Doutor Heitor Vieira Dourado, da Fundação de Vigilância em Saúde do Amazonas, da Fiocruz Amazônia e de diversos hospitais associados ao projeto.
Além da nova metodologia para diagnóstico, o projeto prevê a pesquisa dos mecanismos envolvidos nos distúrbios de coagulação sanguínea – entre eles a alteração na capacidade das plaquetas de se agregar – que têm sido associados à COVID-19. Essa parte da investigação é coordenada pelo professor da USP José Carlos Nicolau. O trabalho descrito no artigo também conta com apoio da FAPESP por meio de auxílios concedidos à professora da USP Ester Sabino e aos professores da Unicamp Wagner José Fávaro e Fabio Trindade Maranhão Costa.
O artigo Covid-19 automated diagnosis and risk assessment through Metabolomics and Machine-Learning pode ser lido em www.medrxiv.org/content/10.1101/2020.07.24.20161828v1.